摘要:肉类调理食品方便快捷,营养美味,深受消费者青睐,但其丰富的营养物质容易造成微生物的滋生,影响食品品质和消费者健康。高通量测序技术可以全面、准确的研究肉类调理食品中微生物群落结构,为微生物防控和标准化生产提供理论基础。本文通过均质提取法对肉类调理食品中微生物基因组进行提取,并以16S rRNA可变区V3-V4作为测序片段,分析细菌多样性。实验表明,4种肉类调理食品细菌多样性均较高,菌群结构具有一定的相似性,炸鸡块、骨肉相连和冷冻牛排的优势菌群是Anderseniella属,而羊肉串优势菌群是不动杆菌属,此外肉类调理食品中还存在芽胞杆菌属、梭菌属、泛菌属、假单胞菌属、嗜冷杆菌属和乳球菌属等。本论文明确了使用均质提取法对肉类调理食品中微生物进行16S rRNA V3-V4区测序分析,为后续大规模菌群研究提供思路和方法。
关键词:肉类调理食品;细菌多样性;高通量测序;16S rRNA
肉类调理食品一般是指以畜和禽肉为主要原料,经适当加工,在冷冻或冷藏条件下贮存、流通和售卖,可直接食用或简单加工后食用的肉类制品,包括炸鸡块、烤翅、羊肉串、肉丸、烟熏火腿和冷冻牛排等。肉类调理食品营养丰富,且由于加工过程中杀菌不彻底,以及我国冷链设备不完善,容易滋生微生物。因此,开展肉类调理食品菌群结构研究,分析微生物多样性,有利于抑制致病菌和腐败菌的产生,对肉类调理食品的质量控制和标准化生产具有一定的指导意义。
在菌群结构的分析检测方面,分子生物学技术,如变性梯度凝胶电泳(DGGE)和限制性片段长度多态性(RFLP)等,相对于传统分离培养法,操作简单,准确度高,但是无法检测样本中低丰度的物种。近年来,新兴的高通量测序技术可以进行大规模平行测序,能够客观反映样本中低丰度和不可培养的细菌,逐渐发展为菌群结构研究的主流技术,广泛应用于医药、环境、食品等各个领域。Abdul-Mutalib等利用焦磷酸测序研究了不同卫生等级餐厅案板上微生物菌群分布情况,发现案板上的菌群多样性较高,且微生物数量与餐厅卫生等级无关,不同卫生等级的餐厅均存在一定的安全隐患。
二代测序以454和Illumina平台应用最为广泛,菌群分析的测序基因主要是16S rRNA的9个可变区,不同可变区对于不同菌属细菌的分辨能力不同,根据研究需要和测序平台选择合适的测序片段,菌群分析相关文献中涉及到的基因片段包括一个可变区(V3、V4和V6)、两个可变区(V1-V2、V3-V4和V4-V5)或三个可变区(V1-V3、V3-V5和V4-V6)等,但目前还没有统一的测序标准。
为了研究肉类调理食品中细菌多样性,本文采用均质提取法对样本中微生物基因组进行提取,利用Illumina MiSeq测序平台完成微生物16S rRNA可变区V3-V4测序,分析肉类调理食品菌群结构,对产品工艺改进和微生物控制提供基础数据。此外,本文明确了使用均质提取法和16S rRNA V3-V4区测序分析肉类调理食品中微生物多样性,为后续大规模菌群研究提供思路和方法。
1 材料与方法
1.1 实验材料
炸鸡块、羊肉串、骨肉相连和冷冻牛排购于北京市某大型超市,样品均为普通袋装,执行速冻调理食品行业标准SB/T 10379-2012,购买后按照产品贮存条件(-18℃以下)进行冷冻保存,在保质期内对样本进行检测。样本信息见表1。
表1 样品主要信息
| 名称 | 规格 | 产地 | 生产日期 |
| 炸鸡块 | 500 g | 山东 | 20150421 |
| 羊肉串 | 240 g | 内蒙古 | 20150128 |
| 骨肉相连 | 400 g | 内蒙古 | 20150422 |
| 牛排 | 300 g | 河北 | 20150506 |
1.2 仪器与设备
拍打式均质机;恒温水浴锅;涡旋振荡器;高速冷冻离心机;小型台式离心机;PCR扩增仪;电泳仪;凝胶成像仪。
1.3 试验方法
1.3.1 样本前处理
以无菌操作取样品25g,充分剪碎后,加入到含有225mL生理盐水的均质袋中,使用拍击式均质器均质1min,获得样品匀液。吸取上清30mL、2000r/min离心10min,沉淀食品杂质;吸取上清液20mL,10000r/min离心3min,缓慢倒掉上清液,获得菌体沉淀。每个样本均设置2个平行,炸鸡块、羊肉串、骨肉相连和冷冻牛排样本编号分别为 J-JK、J-YR、J-GR和J-NP。
1.3.2 基因组DNA提取
使用QIAamp DNA Stool Mini Kit ,根据试剂盒说明书提取菌体沉淀基因组DNA。提取后进行1%琼脂糖凝胶电泳检测提取效果。
1.3.3 高通量测序
提取的基因组DNA 送至生工生物工程(上海)有限公司进行Illumina MiSeq高通量测序。首先使用细菌的通用引物341F(barcode-CCTACGGGNGGCW GCAG)和805R(barcode-GACTACHVGGGTATCTA ATCC)扩增16S rRNA 基因V3-V4区,第二轮扩增引入 Illumina 桥式扩增引物,并利用胶回收纯化扩增产物,经Qubit 2.0精确定量后上机测序。
1.3.4 数据分析
通过Barcode区分样本,并使用FLASH软件将双端测序序列拼接成单条序列。利用Prinseq过滤低质量的序列,完成质量控制。随后使用Mothur软件对序列进行测序错误校正,并采用chimeras.uchime去除序列中的嵌合体,获得优化序列。基于上述优化序列,利用Uclust软件聚类,以序列之间的相似性(0.97作为阈值,分成操作分类单元(Operational Taxonomic Units,OUT),并采用RDP classifier完成物种分类。最后使用R软件进行α多样性指数的计算,绘制菌群丰度图和样本聚类图。
2 结果与讨论
2.1 测序数据信息
本研究使用二代测序技术对微生物16S rRNA V3-V4区进行扩增,测序平均序列读长459 bp,优化后序列平均读长为420bp,通过对序列进行归类操作,按照序列间的距离进行聚类, 以0.97作为序列相似性 阈值划分操作分类单元 (OTU)。 本次实验将所有OTU比对到SILVA数据库,共得到28个门、57个纲、107个目和229个科共783个属,其中包括62个无法鉴定的属。各样本的原始序列数、优化序列数和 OTU 数及在门和属水平对应的细菌数见表 2。由表2可知,测序数据量基本满足分析需求,4 种肉类调理食品中微生物种类很多,平均每个样本中存在三百余种细菌。
表2 样本16S rRNA V3-V4区测序数据信息
| 样品 | 原始序列数 | 优化序列数 | OTU数 | 门个数 | 属个数 |
| J-JK1 | 14394 | 14033 | 2325 | 17 | 296 |
| J-JK2 | 15877 | 15481 | 2671 | 23 | 324 |
| J-YR1 | 15243 | 14407 | 2953 | 17 | 285 |
| J-YR2 | 19105 | 18224 | 3550 | 20 | 322 |
| J-GR1 | 17598 | 17182 | 3143 | 18 | 324 |
| J-GR2 | 21807 | 21289 | 3270 | 20 | 337 |
| J-NP1 | 15331 | 14456 | 2897 | 23 | 347 |
| J-NP2 | 11086 | 10564 | 2402 | 21 | 339 |
Nam等对发酵食品中微生物V1-V2区进行测序分析,样本序列数在18000~25000之间,OTU数在700~1100之间,本研究获得的序列数和OTU数分别在11000~22000和2300~3500之间,样本中细菌菌属在285~347之间,说明实验采用的V3~V4区可以较好地鉴定各类细菌,基本满足物种在属水平上的比较分析。而利用PCR-DGGE技术检测骨肉相连,仅发现假单胞菌属等7个优势菌属,与本实验检出的300余种菌相比,PCR-DGGE技术无法客观反应低丰度物种,容易对微生物物种组成和丰度做出片面的判断。
2.2 α多样性指数
α多样性指某个生境内的多样性,是评价生境内部物种多样性的指标。α多样性主要关注两方面信息,一是群落所含物种种类的多少,即物种丰富度,用Chao1 Index和ACE Index计算;二是群落中各物种的相对数量,即物种均匀度,既考察物种多样性,又考察种的多度, 由Shannon Index和Simpson Index反映。表3显示的是各样本α多样性指数,由表3可知,各样本菌群丰度很高,菌群多样性也较高。测序深度指数(Coverage)是指各样品文库的覆盖率,数值越高,越能反应样本的真实情况,本次实验覆盖率在0.87~0.91之间,基本可以涵盖样本中大多数菌群,测序数据量满足分析的需要。
表3 各样本菌群多样性指数
| 样品 | Chao1 Index | ACE Index | Shannon Index | Simpson Index | Coverage |
| J-JK1 | 4122.26 | 5268.99 | 5.52 | 0.06 | 0.91 |
| J-JK2 | 5127.11 | 7021.04 | 5.38 | 0.05 | 0.90 |
| J-YR1 | 7202.15 | 11397.51 | 6.17 | 0.01 | 0.87 |
| J-YR2 | 7568.19 | 12374.41 | 6.21 | 0.01 | 0.88 |
| J-GR1 | 6643.25 | 10471.15 | 6.00 | 0.02 | 0.89 |
| J-GR2 | 6843.29 | 10479.30 | 5.82 | 0.02 | 0.91 |
| J-NP1 | 5504.03 | 7421.04 | 5.83 | 0.04 | 0.89 |
| J-NP2 | 4672.62 | 6693.76 | 6.00 | 0.02 | 0.87 |
2.3 肉类调理食品细菌多样性
实验样本购买于同一超市,均贮藏于-18℃以下冷冻条件下,但是不同样本的肉类品种来源、加工工艺与生产环境存在差异,因此,各样本之间的主要细菌类群、菌群比例也会存在一定的差异。
在门分类水平上,4 种肉类调理食品中微生物主要是变形菌门和厚壁菌门,这两类细菌占总菌数的80%~90%左右,此外还存在少量的拟杆菌门和放线菌门等。在属水平上,4种肉类调理食品表现出一定的差异,图1显示了各样本属水平物种丰度较高的前20个序列信息。其中,炸鸡块中优势菌群是Anderseniella属(占总序列数的45.21%),此外占总序列数2%以上的还包括乳球菌属(Lactococcus)、明串珠菌属(Leuconostoc)、不动杆菌属(Acinetobacter)、沙雷氏菌属(Serratia)以及未分类的菌属等;羊肉串优势菌群是不动杆菌属(Acinetobacter,占总序列数的26.90%),还存在Anderseniella属、梭菌属(Clostridium)、嗜冷杆菌属(Psychrobacter)、金黄杆菌属(Chryseobacterium)、漫游球菌属(Vagococcus)、乳球菌属(Lactococcus)、假单胞菌属(Pseudomonas)、肠杆菌属(Enterobacter)以及泛菌属 (Pantoea) 等;骨肉相连优势菌群是Anderseniella 属(占总序列数的25.32%),此外还存在泛菌属(Pantoea)、梭菌属(Clostridium)、不动杆菌属(Acinetobacter)、假单胞菌属(Pseudomonas)、嗜冷杆菌属(Psychrobacter)、金黄杆菌属(Chryseobacterium)、香味菌属(Myroides)以及肠杆菌属(Enterobacter)等;冷冻牛排优势菌群是Anderseniella 属(占总序列数的28.49%),此外还存在漫游球菌属(Vagococcus)、肠球菌属(Enterococcus)、假单胞菌属(Pseudomonas)、泛菌属(Pantoea)、链球菌属(Streptococcus)、魏斯氏菌属(Weissel la)以及梭菌属(Clostridium)等。
本实验在肉类调理食品中检出Anderseniella的存在,且占据较大比例。Anderseniella是革兰氏阴性菌,异养需氧型,属于变形菌门、α变形菌纲、根瘤菌目、红菌科,与红菌属(Rhodobium)相似度很高,Anderseniella目前仅包含Anderseniella baltica一个种,曾从波罗的海缺氧表沉积物中分离出来,可以在盐度0.8%~6%之间生长。有关Anderseniella的研究文献较少,目前还没有文献显示Anderseniella在肉类食品中检测或者分离出来。而本实验使用二代测序技术检出Anderseniella在肉类调理食品中存在,这可能与食品的生产工艺有关。炸鸡块、羊肉串、骨肉相连和冷冻牛排生产工艺流程有一定的相似性,将肉类原料预处理(清洗并切块等)后,再加入食盐等调味料腌制一定的时间,熟制或不经过熟制后,真空包装、冷冻保藏。由于调理食品需要进行腌制,腌制过程中高盐度对微生物进行了选择和富集,4种肉类调理食品营养成分表显示,钠含量在4.81mg/g~14mg/g之间,若钠均以氯化钠形式存在,那么换算成盐度,4种食品盐度介于1.22%~3.56%之间,适于Anderseniella的生长,而对其他菌属具有抑制作用,因此腌制过程结束后Anderseniella可能大量存在于食品中。
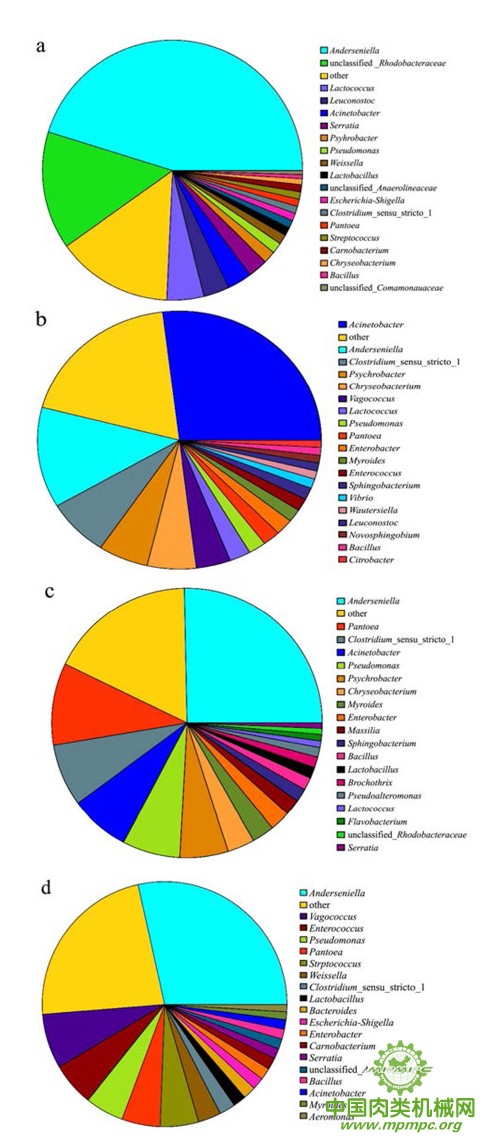
图1 各样本在属水平物种丰度图
注:a,炸鸡块,b,羊肉串;c,骨肉相连;d,牛排。
虽然肉类调理食品种类不同,但菌群结构存在一定的相似性。在物种丰度较高的前20 个序列中,Anderseniella属、不动杆菌属、芽胞杆菌属、梭菌属、泛菌属和假单胞菌属是4种肉类调理食品中的共有菌群,乳球菌属、嗜冷杆菌属、乳杆菌属、香味菌属、漫游球菌属、沙雷氏菌属和泛菌属等也广泛存在于肉类调理食品中。除了Anderseniella,其他菌属在肉及肉制品中常有检出。其中革兰氏阳性菌有乳球菌属、乳杆菌属、芽胞杆菌属、梭菌属和漫游球菌属;革兰氏阴性菌有Anderseniella、假单胞菌属、不动杆菌属、嗜冷杆菌属、泛菌属、香味菌属和沙雷氏菌属。乳球菌属和乳杆菌属是乳品发酵中有益微生物,常用于干酪和酸牛乳的生产;芽胞杆菌属、假单胞菌属、沙雷氏菌属和不动杆菌属广泛存在于自然界,部分菌种是食品腐败菌或人致病菌,如蜡样芽胞杆菌、炭疽芽胞杆菌、铜绿假单胞菌、粘质雷氏菌和鲍曼不动杆菌等。
肉类调理食品中细菌种类较多,除了来源于动物肠道,还可能来源于动物屠宰、食品加工和包装等每一个生产环节,肉及肉制品所接触的外界环境、加工用具、包装材料和人员等均会造成微生物污染,肉制品之间也存在交叉污染。因此,各食品企业需要加强 对原料肉的检测,实施规范化生产、标准化管理,重点对动物屠宰、切割及修整过程微生物污染进行监管和控制,保证产品质量。此外,很多肉类调理食品在流通和贮藏过程中需要冷藏或冷冻保存,而我国冷链系统尚不完备,温度的波动也为微生物的生长繁殖提供条件。
值得关注的是,肉类调理食品中的优势菌群并不是嗜冷菌,这与我们实验前的理论推测是不同的。实验结果表明,尽管样本的贮存条件是-18℃,但仍然存在生长温度在30℃~37℃的中温菌,如不动杆菌属、梭菌属、泛菌属和明串珠菌属等。这些菌属很可能处于“活的不可培养状态(Viable but nonculturable state,VBNC)”,即丧失细菌可培养的特征,但是细胞膜完整,有一定的代谢活力,遇到适宜条件,可恢复到可培养的状态,这是某些无法产生芽胞的细菌对抗环境压力的一种反应。研究表明,温度是诱导细菌进入VBNC状态最主要的原因,因此肉类调理食品在冷冻处理时,埃希氏菌属、志贺氏菌、肠球菌和弧菌等很可能进入VBNC状态,一旦遇到适宜的生长条件,便会再次复苏,对消费者的身体健康造成危害。
2.4 样本间菌群结构比较
维恩图可在OTU水平对4种肉类调理食品中菌群进行分析,图2直观的反映了样本间的共有OTU和特有OTU。为了清楚显示,本实验将2个平行样本合并分析(J-JK1和J-JK2合并为J-JK,其他样本类同),J-JK、J-YR、J-GR和J-NP获得的OTU数分别为3486、5014、5122和3773,所有样本共含有12000余种不同的OTU。由图可知,每2种实验样本之间相同的OTU数在1019~1902之间,4种样本之间相同的OTU数为502个,占总OTU数的4.16%。
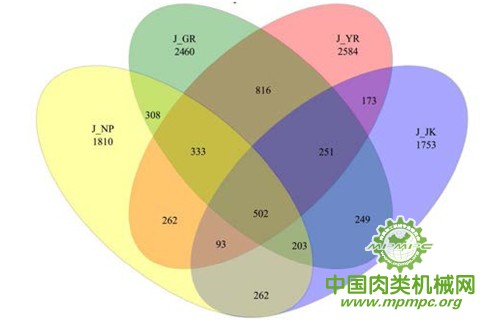
图2 操作分类单元维恩图
为了从微生物种类和丰度层面研究4种肉类调理食品的异同,本研究基于Bray-Curtis指数绘制样本聚类图,如图3所示,菌群组成相似度越高的样本在图中的距离越小。整体而言,4 种肉类调理食品之间具有很大的相似性,其中,炸鸡块和牛排、羊肉串和骨肉相连相似度较高,分别聚为一簇。而同为鸡肉制品的炸鸡块和骨肉相连并未表现出明显的相似性,这可能是二者加工工艺、加工环境不同引起的。此外,在样品中检测出了不可分类微生物,如unclassified Rhodobacteraceae和unclassified Anaerolineaceae等,这是由于测序结果与数据库中序列不匹配或相似度不够阈值,因此这些序列被归为不可分类的微生物。
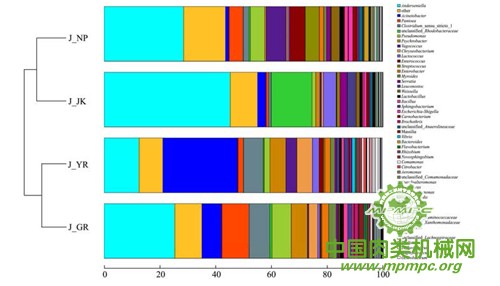
图3 各样本在属水平聚类图
3 结论
利用高通量测序技术可以直接从食品中提取微生物基因组,对不可培养细菌和低丰度细菌也可有效检测,能够客观分析食品中菌群结构,且高通量测序技术可以对多个样本同时检测,相对于传统方法能够迅速、高效的完成样本菌群分析。本论文明确了使用均质提取法提取微生物基因组,并采用二代测序技术进行16S rRNA V3-V4区测序,为后续大规模菌群研究提供思路和方法。实验结果表明,肉类调理食品中细菌多样性高,4种肉类调理食品菌群结构具有一定的相似性,在门分类水平,主要是变形菌门和厚壁菌门组成;在属水平,Anderseniella 属、不动杆菌属、芽胞杆菌属、嗜冷杆菌属、乳球菌属、梭菌属、泛菌属和假单胞菌属等广泛存在,这些微生物可能来源于动物肠道、动物屠宰、食品加工和包装等加工工序,因此,各食品企业需要优化生产工艺,加强生产管理,制定标准化操作流程,保证产品质量稳定。








